R语言缺失值处理
缺失数据的分类:
完全随机缺失:若某变量的缺失数据与其他任何观测或未观测变量都不相关,则数据为完全随机缺失(MCAR)。
随机缺失:若某变量上的缺失数据与其他观测变量相关,与它自己的未观测值不相关,则数据为随机缺失(MAR)。
非随机缺失:若缺失数据不属于MCAR或MAR,则数据为非随机缺失(NMAR) 。
处理缺失数据的方法有很多,但哪种最适合你,需要在实践中检验。
下面一副图形展示处理缺失数据的方法:
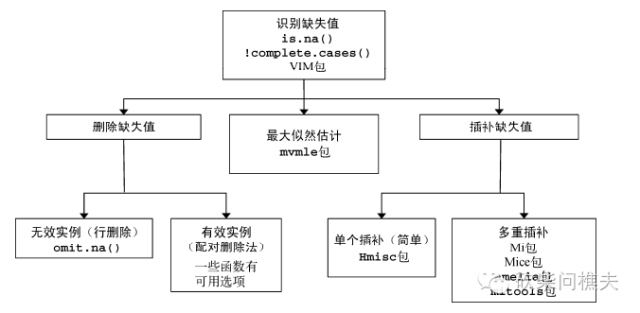
处理数据缺失的一般步骤:
1、识别缺失数据
2、检测导致数据缺失的原因
3、删除包含缺失值的实例或用合理的数值代替(插补)缺失值。
1、识别缺失数据:
R语言中,NA代表缺失值,NaN代表不可能值,Inf和-Inf代表正无穷和负无穷。
在这里,推荐使用is.na,is.nan,is.finite,is.infinite4个函数去处理。
complete.case() 可用来识别矩阵或数据框中没有缺失值的行
> library(VIM)
> data(sleep)
> View(sleep)
> sleep[!complete.cases(sleep),]
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
1 6654.000 5712.0 NA NA 3.3 38.6 645 3 5 3
3 3.385 44.5 NA NA 12.5 14.0 60 1 1 1
4 0.920 5.7 NA NA 16.5 NA 25 5 2 3
13 0.550 2.4 7.6 2.7 10.3 NA NA 2 1 2
14 187.100 419.0 NA NA 3.1 40.0 365 5 5 5
19 1.410 17.5 4.8 1.3 6.1 34.0 NA 1 2 1
20 60.000 81.0 12.0 6.1 18.1 7.0 NA 1 1 1
21 529.000 680.0 NA 0.3 NA 28.0 400 5 5 5
24 207.000 406.0 NA NA 12.0 39.3 252 1 4 1
26 36.330 119.5 NA NA 13.0 16.2 63 1 1 1
30 100.000 157.0 NA NA 10.8 22.4 100 1 1 1
31 35.000 56.0 NA NA NA 16.3 33 3 5 4
35 0.122 3.0 8.2 2.4 10.6 NA 30 2 1 1
36 1.350 8.1 8.4 2.8 11.2 NA 45 3 1 3
41 250.000 490.0 NA 1.0 NA 23.6 440 5 5 5
47 4.288 39.2 NA NA 12.5 13.7 63 2 2 2
53 14.830 98.2 NA NA 2.6 17.0 150 5 5 5
55 1.400 12.5 NA NA 11.0 12.7 90 2 2 2
56 0.060 1.0 8.1 2.2 10.3 3.5 NA 3 1 2
62 4.050 17.0 NA NA NA 13.0 38 3 1 1
判断数据有多少缺失值
> sum(is.na(sleep))
[1] 38
针对复杂的数据集,怎么更好的探索数据缺失情况呢?
mice包 中的 md.pattern() 函数可以生成一个以矩阵或数据框形式展示缺失值模式的表格。
> library(mice)
> md.pattern(sleep)
BodyWgt BrainWgt Pred Exp Danger Sleep Span Gest Dream NonD
42 1 1 1 1 1 1 1 1 1 1 0
2 1 1 1 1 1 1 0 1 1 1 1
3 1 1 1 1 1 1 1 0 1 1 1
9 1 1 1 1 1 1 1 1 0 0 2
2 1 1 1 1 1 0 1 1 1 0 2
1 1 1 1 1 1 1 0 0 1 1 2
2 1 1 1 1 1 0 1 1 0 0 3
1 1 1 1 1 1 1 0 1 0 0 3
0 0 0 0 0 4 4 4 12 14 38备注:0表示变量的列中没有缺失,1则表示有缺失值。
第一行给出了没有缺失值的数目(共多少行)。
第一列表示各缺失值的模式。
最后一行给出了每个变量的缺失值数目。
最后一列给出了变量的数目(这些变量存在缺失值)。
在这个数据集中,总共有38个数据缺失。
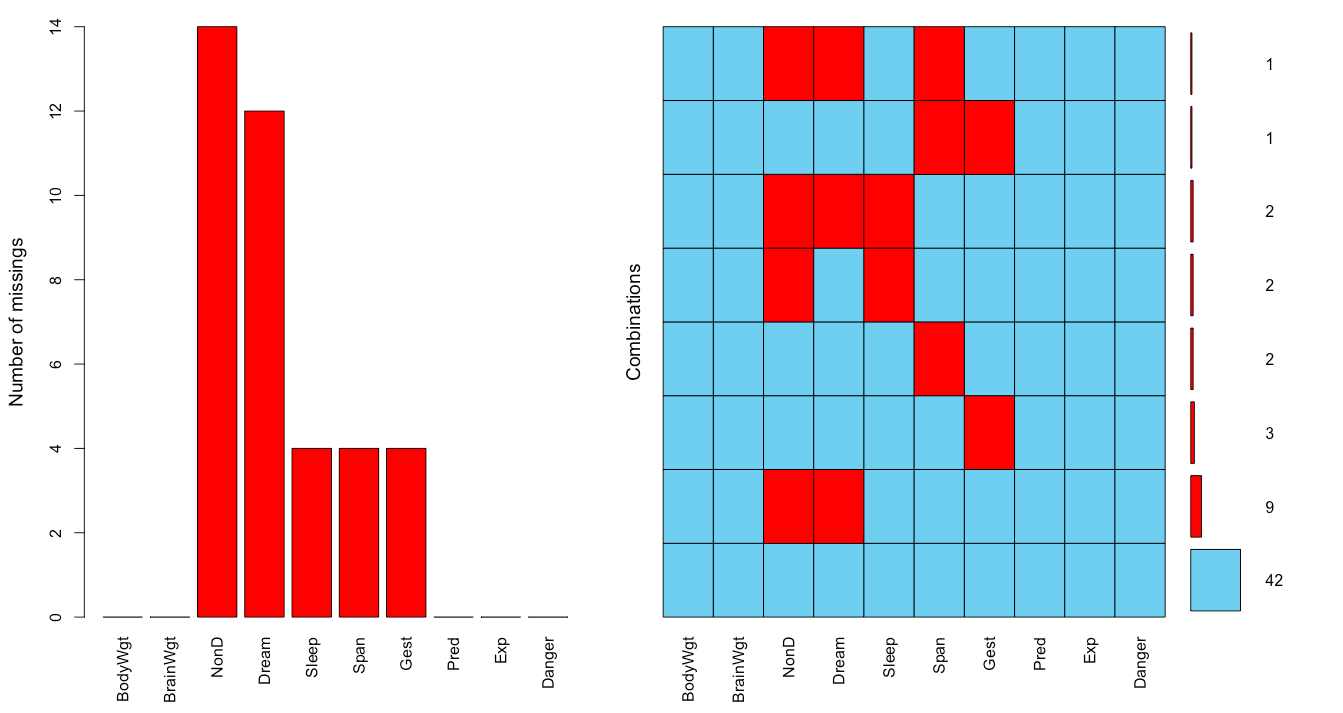
图形化展示缺失数据:
> aggr(sleep,prop=F,numbers=T)
> matrixplot(sleep)
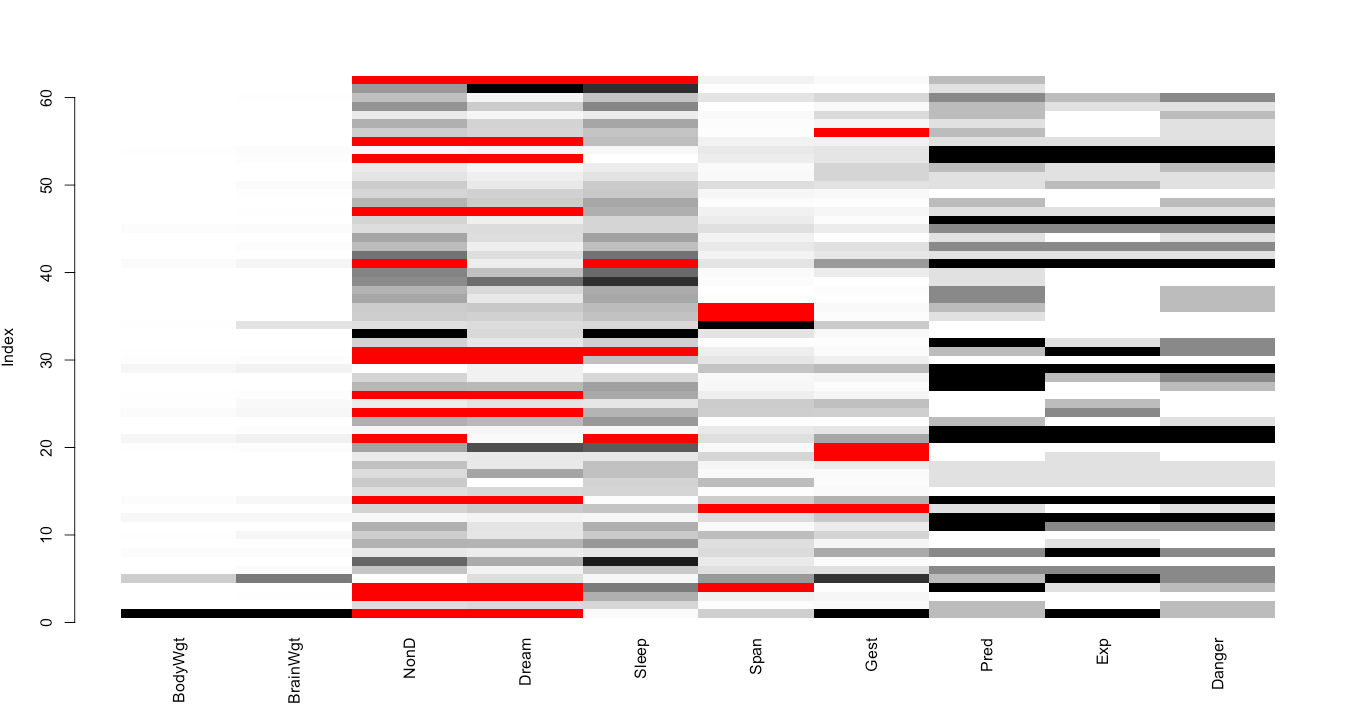
浅色表示值小,深色表示值大,默认缺失值为红色。
2、缺失值数据的处理
行删除法:数据集中含有缺失值的行都会被删除,一般假定缺失数据是完全随机产生的,并且缺失值只是很少一部分,对结果不会造成大的影响。
即:要有足够的样本量,并且删除缺失值后不会有大的偏差!
行删除的函数有na.omit()和complete.case()
newdata<-na.omit(sleep)
newdata<-sleep[complete.cases(sleep),]
均值/中位数等填充:这种方法简单粗暴,如果填充值对结果影响不怎么大,这种方法倒是可以接受,并且有可能会产生令人满意的结果。
Hmisc包 更加简单,可以插补均值、中位数等,你也可以插补指定值。
> library(Hmisc)
> newdata<-sleep
> impute(newdata$Dream,mean)
> impute(newdata$Dream,median)
> impute(newdata$Dream,2)
mice包插补缺失数据: 链式方程多元插值,首先利用mice函数建模再用complete函数生成完整数据。
下图展示mice包的操作过程:
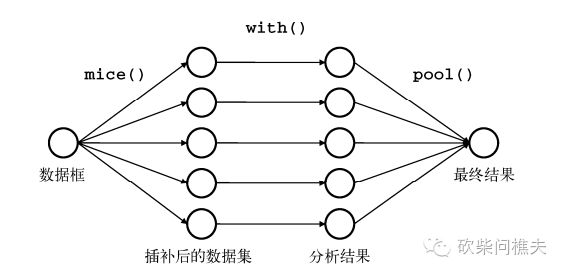
mice():从一个含缺失值的数据框开始,返回一个包含多个完整数据集对象(默认可以模拟参数5个完整的数据集)
with():可依次对每个完整数据集应用统计建模
pool():将with()生成的单独结果整合到一起
> library(mice)
> newdata<-sleep
> data<-mice(newdata,m=5,method = "pmm",maxit = 100,seed=1)插补方法是pmm:预测均值匹配,可以用methods(mice)查看其他方法
maxit指迭代次数,seed指设定种子数(和set.seed同义)
> methods(mice)
[1] mice.impute.2l.norm mice.impute.2l.pan mice.impute.2lonly.mean
[4] mice.impute.2lonly.norm mice.impute.2lonly.pmm mice.impute.cart
[7] mice.impute.fastpmm mice.impute.lda mice.impute.logreg
[10] mice.impute.logreg.boot mice.impute.mean mice.impute.midastouch
[13] mice.impute.norm mice.impute.norm.boot mice.impute.norm.nob
[16] mice.impute.norm.predict mice.impute.passive mice.impute.pmm
[19] mice.impute.polr mice.impute.polyreg mice.impute.quadratic
[22] mice.impute.rf mice.impute.ri mice.impute.sample
[25] mice.mids mice.theme
see '?methods' for accessing help and source code
> summary(data)
Multiply imputed data set
Call:
mice(data = newdata, m = 5, method = "pmm", maxit = 100, seed = 1)
Number of multiple imputations: 5
Missing cells per column:
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
0 0 14 12 4 4 4 0 0 0
Imputation methods:
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
"pmm" "pmm" "pmm" "pmm" "pmm" "pmm" "pmm" "pmm" "pmm" "pmm"
VisitSequence:
NonD Dream Sleep Span Gest
3 4 5 6 7
PredictorMatrix:
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
BodyWgt 0 0 0 0 0 0 0 0 0 0
BrainWgt 0 0 0 0 0 0 0 0 0 0
NonD 1 1 0 1 1 1 1 1 1 1
Dream 1 1 1 0 1 1 1 1 1 1
Sleep 1 1 1 1 0 1 1 1 1 1
Span 1 1 1 1 1 0 1 1 1 1
Gest 1 1 1 1 1 1 0 1 1 1
Pred 0 0 0 0 0 0 0 0 0 0
Exp 0 0 0 0 0 0 0 0 0 0
Danger 0 0 0 0 0 0 0 0 0 0
Random generator seed value: 1 在这上面可以看到数据集中变量的观测值缺失情况,每个变量的插补方法, VisitSequence 从左至右展示了插补的变量, 预测变量矩阵 (PredictorMatrix)展示了进行插补过程的含有缺失数据的变量,它们利用了数据集中其他变量的信息。(在矩阵中,行代表插补变量,列代表为插补提供信息的变量, 1和0分别表示使用和未使用。)
查看整体插补的数据:
> data$imp查看具体变量的插补数据:
> data$imp$Dream最后,最重要的是生成一个完整的数据集
> completedata<-complete(data)
> head(completedata)
BodyWgt BrainWgt NonD Dream Sleep Span Gest Pred Exp Danger
1 6654.000 5712.0 2.1 0.5 3.3 38.6 645 3 5 3
2 1.000 6.6 6.3 2.0 8.3 4.5 42 3 1 3
3 3.385 44.5 10.9 1.8 12.5 14.0 60 1 1 1
4 0.920 5.7 13.2 3.1 16.5 2.3 25 5 2 3
5 2547.000 4603.0 2.1 1.8 3.9 69.0 624 3 5 4
6 10.550 179.5 9.1 0.7 9.8 27.0 180 4 4 4判断还有没有缺失值,如果没有,结果返回FLASE
> anyNA(completedata)
针对以上插补结果,我们可以查看原始数据和插补后的数据的分布情况
图上,插补值是洋红点呈现出的形状,观测值是蓝色点。
> densityplot(data)
图上,洋红线是每个插补数据集的数据密度曲线,蓝色是观测值数据的密度曲线
> stripplot(data,pch=12)
上图中,0代表原始数据,1-5代表5次插补的数据,洋红色的点代表插补值
下面我们分析对数据拟合一个线性模型:
完整数据:
> library(mice)
> newdata<-sleep
> data<-mice(newdata,m = 5,method=’pmm’,maxit=100,seed=1)
> model<-with(data,lm(Dream~Span+Gest))
> pooled<-pool(model)
> summary(pooled)
est se t df Pr(>|t|) lo 95
(Intercept) 2.527581179 0.24068442 10.5016403 54.36478 1.065814e-14 2.045112117
Span -0.005492745 0.01139327 -0.4821045 55.44497 6.316294e-01 -0.028321243
Gest -0.003754241 0.00142867 -2.6277875 53.85804 1.117229e-02 -0.006618725
hi 95 nmis fmi lambda
(Intercept) 3.0100502401 NA 0.06699282 0.03328882
Span 0.0173357526 4 0.05569090 0.02223144
Gest -0.0008897574 4 0.07184834 0.03801003
fim指的是各个变量缺失信息的比例,lambda指的是每个变量对缺失数据的贡献大小
缺失数据(在运行中,自动会行删除):
> lm.fit <- lm(Dream~Span+Gest, data = sleep,na.action=na.omit)

